Python3 数据科学(三):Pandas
Pandas 入门
与 numpy 库专门处理矩阵运算不同的是 pandas 是一个进行数据处理的库。pandas 在 numpy 的基础上进行了相应的封装,专门用来处理数据,进行数据的预处理操作。

导入 Pandas
import pandas as pd# 标准写法
这是导入 pandas 的标准方法。我们不想一直写 pandas 的全名,但是保证代码的简洁和避免命名冲突都很重要,所以折中使用 pd 。如果你去看别人使用 pandas 的代码,就会看到这种导入方式。
Pandas 中的数据类型
Pandas 基于两种数据类型,series 和 dataframe。
series 是一种一维的数据类型,其中的每个元素都有各自的标签。可以把它当作一个由带标签的元素组成的 numpy 数组。标签可以是数字或者字符。
dataframe 是一个二维的、表格型的数据结构。Pandas 的 dataframe 可以储存许多不同类型的数据,并且每个轴都有标签。你可以把它当作一个 series 的字典。
将数据导入 Pandas
在对数据进行修改、探索和分析之前,我们得先导入数据。
这里我鼓励你去找到自己感兴趣的数据并用来练习,一些公开的数据集可以在 Kaggle 上找到。
这里我使用的是食物营养信息数据集,本文所用到的数据下载地址
food_info = pd.read_csv('data/food_info.csv')
这里我们从 csv 文件里导入了数据,并储存在 dataframe 中。这一步非常简单,你只需要调用 read_csv 然后将文件的路径传进去就行了。
探索和分析数据
现在数据已经导入到 Pandas 了,我们也许想看一眼数据来得到一些基本信息,以便在真正开始探索之前找到一些方向。
查看前 x 行的数据:
food_info.head()
我们只需要调用 head() 函数并且将想要查看的行数传入。默认显示的行数为5
你可能还想看看最后几行:
food_info.tail()
跟 head 一样,我们只需要调用 tail 并且传入想要查看的行数即可。默认为5行
注意,它并不是从最后一行倒着显示的,而是按照数据原来的顺序显示。
我们已经看到了数据的前几行和后几行,这些数据都有相同的36列属性,我们想看下这些属性的具体信息便可以使用下面的方式:
food_info.dtypes
NDB_No int64
Shrt_Desc object
Water_(g) float64
Energ_Kcal int64
Protein_(g) float64
Lipid_Tot_(g) float64
Ash_(g) float64
Carbohydrt_(g) float64
Fiber_TD_(g) float64
Sugar_Tot_(g) float64
Calcium_(mg) float64
Iron_(mg) float64
Magnesium_(mg) float64
Phosphorus_(mg) float64
Potassium_(mg) float64
Sodium_(mg) float64
Zinc_(mg) float64
Copper_(mg) float64
Manganese_(mg) float64
Selenium_(mcg) float64
Vit_C_(mg) float64
Thiamin_(mg) float64
Riboflavin_(mg) float64
Niacin_(mg) float64
Vit_B6_(mg) float64
Vit_B12_(mcg) float64
Vit_A_IU float64
Vit_A_RAE float64
Vit_E_(mg) float64
Vit_D_mcg float64
Vit_D_IU float64
Vit_K_(mcg) float64
FA_Sat_(g) float64
FA_Mono_(g) float64
FA_Poly_(g) float64
Cholestrl_(mg) float64
dtype: object
上面便展示了每一列数据的类型,这些类型所代表的意思为:
- object - 字符串类型
- int - 整数类型
- float - 浮点数类型
- datetime - 时间类型
- bool - bool值类型
想要获取数据所有的列名,可以使用 colimns 属性来获取
food_info.columns.tolist()
['NDB_No',
'Shrt_Desc',
'Water_(g)',
'Energ_Kcal',
'Protein_(g)',
'Lipid_Tot_(g)',
'Ash_(g)',
'Carbohydrt_(g)',
'Fiber_TD_(g)',
'Sugar_Tot_(g)',
'Calcium_(mg)',
'Iron_(mg)',
'Magnesium_(mg)',
'Phosphorus_(mg)',
'Potassium_(mg)',
'Sodium_(mg)',
'Zinc_(mg)',
'Copper_(mg)',
'Manganese_(mg)',
'Selenium_(mcg)',
'Vit_C_(mg)',
'Thiamin_(mg)',
'Riboflavin_(mg)',
'Niacin_(mg)',
'Vit_B6_(mg)',
'Vit_B12_(mcg)',
'Vit_A_IU',
'Vit_A_RAE',
'Vit_E_(mg)',
'Vit_D_mcg',
'Vit_D_IU',
'Vit_K_(mcg)',
'FA_Sat_(g)',
'FA_Mono_(g)',
'FA_Poly_(g)',
'Cholestrl_(mg)']
在日常的使用过程中,获取到数据最常用的便是得看下数据中格式,这里因为 dataframe 格式的数据也是以矩阵的形式展示,因此可以使用 shape 属性来查看:
food_info.shape
(8618, 36)
可以使用 info 方法来查看数据中每一列的数据情况:
food_info.info()
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 8618 entries, 0 to 8617
Data columns (total 36 columns):
NDB_No 8618 non-null int64
Shrt_Desc 8618 non-null object
Water_(g) 8612 non-null float64
Energ_Kcal 8618 non-null int64
Protein_(g) 8618 non-null float64
Lipid_Tot_(g) 8618 non-null float64
Ash_(g) 8286 non-null float64
Carbohydrt_(g) 8618 non-null float64
Fiber_TD_(g) 7962 non-null float64
Sugar_Tot_(g) 6679 non-null float64
Calcium_(mg) 8264 non-null float64
Iron_(mg) 8471 non-null float64
Magnesium_(mg) 7936 non-null float64
Phosphorus_(mg) 8046 non-null float64
Potassium_(mg) 8208 non-null float64
Sodium_(mg) 8535 non-null float64
Zinc_(mg) 7917 non-null float64
Copper_(mg) 7363 non-null float64
Manganese_(mg) 6478 non-null float64
Selenium_(mcg) 6868 non-null float64
Vit_C_(mg) 7826 non-null float64
Thiamin_(mg) 7939 non-null float64
Riboflavin_(mg) 7961 non-null float64
Niacin_(mg) 7937 non-null float64
Vit_B6_(mg) 7677 non-null float64
Vit_B12_(mcg) 7427 non-null float64
Vit_A_IU 7932 non-null float64
Vit_A_RAE 7089 non-null float64
Vit_E_(mg) 5613 non-null float64
Vit_D_mcg 5319 non-null float64
Vit_D_IU 5320 non-null float64
Vit_K_(mcg) 4969 non-null float64
FA_Sat_(g) 8274 non-null float64
FA_Mono_(g) 7947 non-null float64
FA_Poly_(g) 7954 non-null float64
Cholestrl_(mg) 8250 non-null float64
dtypes: float64(33), int64(2), object(1)
memory usage: 2.4+ MB
过滤
在探索数据的时候,你可能经常想要抽取数据中特定的样本,比如你有一个关于工作满意度的调查表,你可能就想要提取特定行业或者年龄的人的数据。
在 Pandas 中有多种方法可以实现提取我们想要的信息:
有时你想提取一整列,使用列的标签可以非常简单地做到:
food_info['Vit_A_IU'].head()
0 2499.0
1 2499.0
2 3069.0
3 721.0
4 1080.0
Name: Vit_A_IU, dtype: float64
注意,当我们提取列的时候,会得到一个 series ,而不是 dataframe 。记得我们前面提到过,你可以把 dataframe 看作是一个 series 的字典,所以在抽取列的时候,我们就会得到一个 series。
还记得我在命名列标签的时候特意指出的吗?不用空格、破折号之类的符号,这样我们就可以像访问对象属性一样访问数据集的列——只用一个点号。
food_info.Vit_A_IU.head()
0 2499.0
1 2499.0
2 3069.0
3 721.0
4 1080.0
Name: Vit_A_IU, dtype: float64
这句代码返回的结果与前一个例子完全一样——是我们选择的那列数据。
也可以获取数据集中的多列数据
food_info[['Zinc_(mg)', 'Copper_(mg)']].head()
如果数据中含有字符串,还可以使用字符串方法来进行过滤。比如,我们来获取数据所有列中以 ‘g’ 结尾的列名:
col_names = food_info.columns.tolist()
gram_columns = []
for c in col_names:
if c.endswith("(g)"):
gram_columns.append(c)
gram_df = food_info[gram_columns]
print(gram_df.head())
Water_(g) Protein_(g) Lipid_Tot_(g) Ash_(g) Carbohydrt_(g) \
0 15.87 0.85 81.11 2.11 0.06
1 15.87 0.85 81.11 2.11 0.06
2 0.24 0.28 99.48 0.00 0.00
3 42.41 21.40 28.74 5.11 2.34
4 41.11 23.24 29.68 3.18 2.79
Fiber_TD_(g) Sugar_Tot_(g) FA_Sat_(g) FA_Mono_(g) FA_Poly_(g)
0 0.0 0.06 51.368 21.021 3.043
1 0.0 0.06 50.489 23.426 3.012
2 0.0 0.00 61.924 28.732 3.694
3 0.0 0.50 18.669 7.778 0.800
4 0.0 0.51 18.764 8.598 0.784
索引
之前的部分展示了如何通过列操作来得到数据,但是 Pandas 的行也有标签。行标签可以是基于位置的或者是标签,而且获取行数据的方法也根据标签的类型各有不同。
对于一个数据集来说,它是按行从第0行开始排序的,这也是它的位置。可以通过 iloc 来引用:
food_info.iloc[0]
NDB_No 1001
Shrt_Desc BUTTER WITH SALT
Water_(g) 15.87
Energ_Kcal 717
Protein_(g) 0.85
Lipid_Tot_(g) 81.11
Ash_(g) 2.11
Carbohydrt_(g) 0.06
Fiber_TD_(g) 0
Sugar_Tot_(g) 0.06
Calcium_(mg) 24
Iron_(mg) 0.02
Magnesium_(mg) 2
Phosphorus_(mg) 24
Potassium_(mg) 24
Sodium_(mg) 643
Zinc_(mg) 0.09
Copper_(mg) 0
Manganese_(mg) 0
Selenium_(mcg) 1
Vit_C_(mg) 0
Thiamin_(mg) 0.005
Riboflavin_(mg) 0.034
Niacin_(mg) 0.042
Vit_B6_(mg) 0.003
Vit_B12_(mcg) 0.17
Vit_A_IU 2499
Vit_A_RAE 684
Vit_E_(mg) 2.32
Vit_D_mcg 1.5
Vit_D_IU 60
Vit_K_(mcg) 7
FA_Sat_(g) 51.368
FA_Mono_(g) 21.021
FA_Poly_(g) 3.043
Cholestrl_(mg) 215
Name: 0, dtype: object
但是如果设置的索引列中是字符型数据,这意味着我们不能继续使用 iloc 位置来引用,那我们用 loc 。
下面的代码将将数据集的索引设置为 Shrt_Desc 新索引:
food_info_1 = food_info.set_index(['Shrt_Desc'])
food_info_1.head()
food_info_1.loc['BUTTER WITH SALT']
NDB_No 1001.000
Water_(g) 15.870
Energ_Kcal 717.000
Protein_(g) 0.850
Lipid_Tot_(g) 81.110
Ash_(g) 2.110
Carbohydrt_(g) 0.060
Fiber_TD_(g) 0.000
Sugar_Tot_(g) 0.060
Calcium_(mg) 24.000
Iron_(mg) 0.020
Magnesium_(mg) 2.000
Phosphorus_(mg) 24.000
Potassium_(mg) 24.000
Sodium_(mg) 643.000
Zinc_(mg) 0.090
Copper_(mg) 0.000
Manganese_(mg) 0.000
Selenium_(mcg) 1.000
Vit_C_(mg) 0.000
Thiamin_(mg) 0.005
Riboflavin_(mg) 0.034
Niacin_(mg) 0.042
Vit_B6_(mg) 0.003
Vit_B12_(mcg) 0.170
Vit_A_IU 2499.000
Vit_A_RAE 684.000
Vit_E_(mg) 2.320
Vit_D_mcg 1.500
Vit_D_IU 60.000
Vit_K_(mcg) 7.000
FA_Sat_(g) 51.368
FA_Mono_(g) 21.021
FA_Poly_(g) 3.043
Cholestrl_(mg) 215.000
Name: BUTTER WITH SALT, dtype: float64
和 iloc 一样,loc 会返回你引用的列,唯一一点不同就是此时你使用的是基于字符串的引用,而不是基于数字的。
还有一个引用列的常用常用方法—— ix 。如果 loc 是基于标签的,而 iloc 是基于位置的,但是 ix 已经被弃用了,故这里不做多的介绍。
将数据进行排序通常会很有用,在 Pandas 中,我们可以对 dataframe 调用 sort_values 方法进行排序。inplace 表示是否将排序后的结果替换原来的列。
food_info.sort_values('Sodium_(mg)', inplace=True)
food_info['Sodium_(mg)'].head()
760 0.0
405 0.0
761 0.0
2269 0.0
763 0.0
Name: Sodium_(mg), dtype: float64
上面这句是默认的升序排序,假如我们想要降序排序的话便可以指定 ascending = False
food_info.sort_values('Sodium_(mg)', inplace=True, ascending=False)
food_info['Sodium_(mg)'].head()
276 38758.0
5814 27360.0
6192 26050.0
1242 26000.0
1245 24000.0
Name: Sodium_(mg), dtype: float64
将索引排序通常会很有用,在 Pandas 中,我们可以对 dataframe 调用 sort_index 方法进行排序。
food_info_2 = food_info.set_index(['NDB_No'])
food_info_2.sort_index(ascending=False).head(5)
我的索引本来就是有序的,为了演示,我将参数 ascending 设置为 false,这样我的数据就会呈降序排列。
当你将一列设置为索引的时候,它就不再是数据的一部分了。如果你想将索引恢复为数据,调用 set_index 相反的方法 reset_index 即可:
food_info_2 = food_info_2.reset_index(['NDB_No'])
food_info_2.head(5)
对数据集应用函数
有时你想对数据集中的数据进行改变或者某种操作。比方说,你的数据集中的数据是以 mg 为单位的,你想创建一个新的列以 g 为单位。Pandas 中有两个非常有用的函数,apply 和 applymap。
def mg_to_g(mg):
return mg/1000
food_info['Cholestrl_(g)'] = food_info['Cholestrl_(mg)'].apply(mg_to_g)
food_info.head()
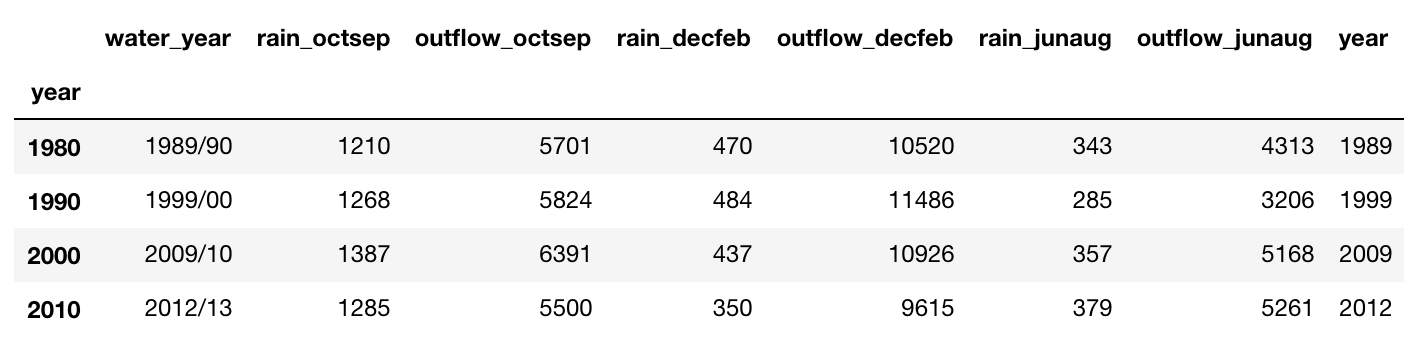
上面的代码创建了一个叫做 Cholestrl_(g) 的列,它只将 Cholestrl_(mg) 列中的数据进行了除1000。这就是 apply 的用法,即对一列数据应用函数。如果你想对整个数据集应用函数,就要使用 applymap 。
操作数据集的结构
另一常见的做法是重新建立数据结构,使得数据集呈现出一种更方便并且(或者)有用的形式。
这里我们会使用一份新的数据集来做下面的测试,数据集为英国2014年的降雨数据:uk_rain_2014.csv
df = pd.read_csv('data/uk_rain_2014.csv')
df.columns = ['water_year','rain_octsep', 'outflow_octsep',
'rain_decfeb', 'outflow_decfeb', 'rain_junaug', 'outflow_junaug']
def base_year(year):
base_year = year[:4]
base_year= pd.to_datetime(base_year).year
return base_year
df['year'] = df['water_year'].apply(base_year)
groupby
groupby 会按照你选择的列对数据集进行分组。
df.groupby(df['year'] // 10 *10).max()
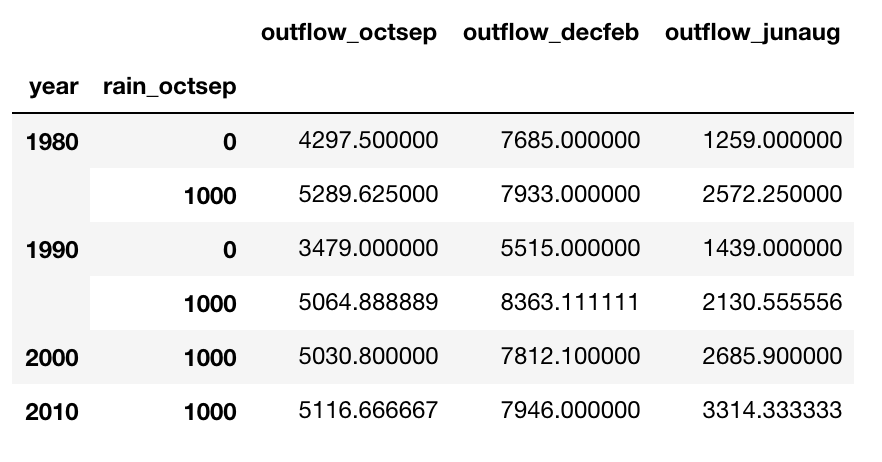
groupby 会按照你选择的列对数据集进行分组。上例是按照年代分组。不过仅仅这样做并没有什么用,我们必须对其调用函数,比如 max 、 min 、mean 等等。例中,我们可以得到 90 年代的均值。
你也可以按照多列进行分组:
decade_rain = df.groupby([df['year'] // 10 * 10, df.rain_octsep // 1000 * 1000])[['outflow_octsep', 'outflow_decfeb', 'outflow_junaug']].mean()
decade_rain
unstack
它可以将一列数据设置为列标签
decade_rain.unstack(0)
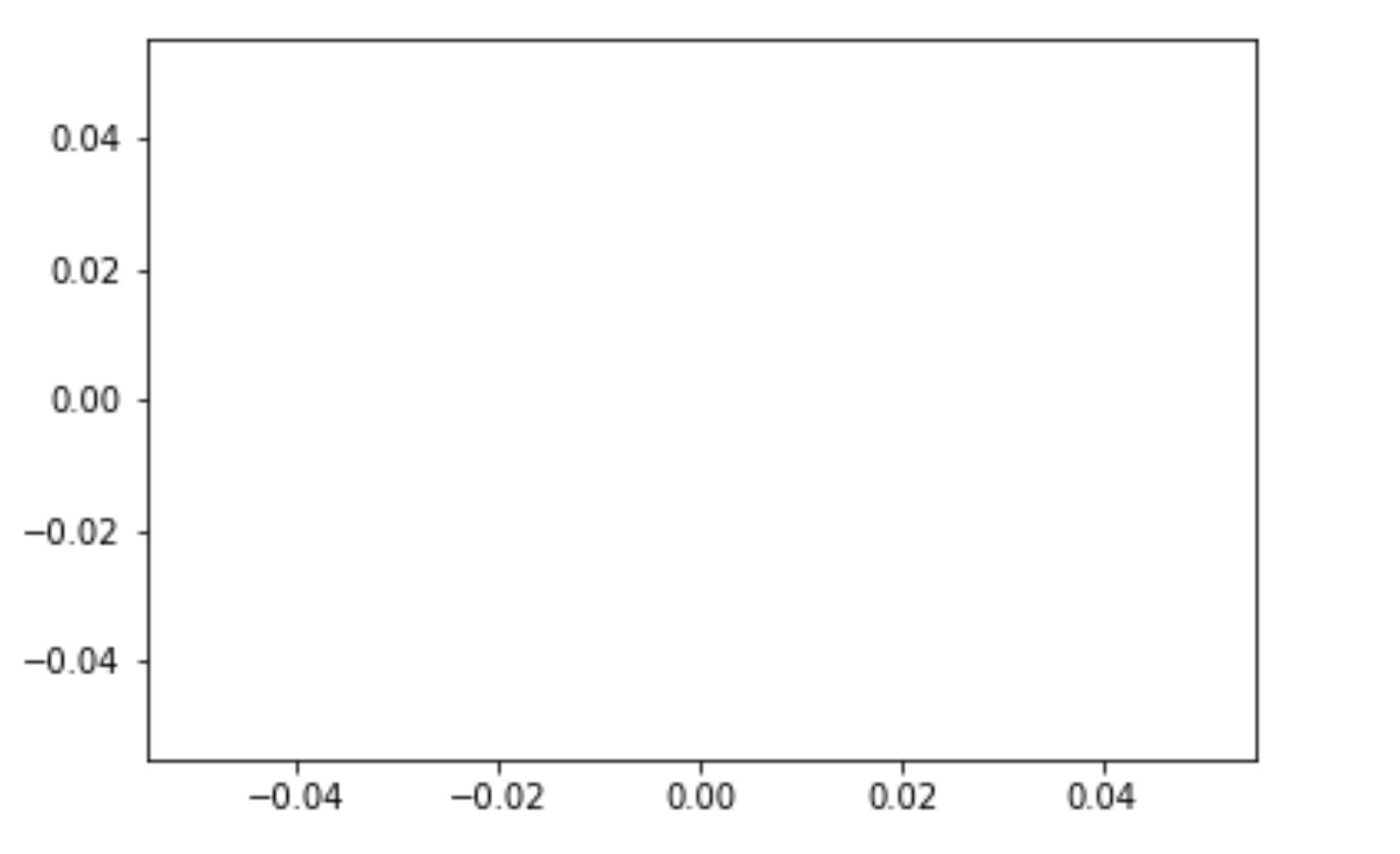
这条语句将上例中的 dataframe 转换为下面的形式。它将第 0 列,也就是 year 列设置为列的标签。
合并数据集
有时你有两个相关联的数据集,你想将它们放在一起比较或者合并它们。在 Pandas 里很简单:
rain_jpn = pd.read_csv('jpn_rain.csv')
rain_jpn.columns = ['year', 'jpn_rainfall']
uk_jpn_rain = df.merge(rain_jpn, on='year')
uk_jpn_rain.head(5)
首先你需要通过 on 关键字来指定需要合并的列。通常你可以省略这个参数,Pandas 将会自动选择要合并的列。
使用 Pandas 快速作图
Matplotlib 很棒,但是想要绘制出还算不错的图表却要写不少代码,而有时你只是想粗略的做个图来探索下数据,搞清楚数据的含义。Pandas 通过 plot 来解决这个问题:
uk_jpn_rain.plot(x='year', y=['rain_octsep', 'jpn_rainfall'])
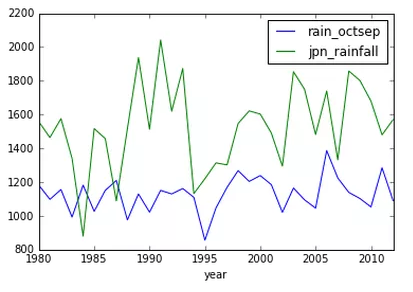
这会调用 Matplotlib 快速轻松地绘出了你的数据图。通过这个图你就可以在视觉上分析数据,而且它能在探索数据的时候给你一些方向。比如,看数据图,你会发现在 1995 年的英国好像有一场干旱。
你会发现英国的降雨明显少于日本,但人们却说英国总是下雨。
保存你的数据集
在清洗、重塑、探索完数据之后,你最后的数据集可能会发生很大改变,并且比最开始的时候更有用。你应该保存原始的数据集,但是你同样应该保存处理之后的数据。
df.to_csv('uk_rain.csv')
上面的代码将会保存你的数据到 csv 文件以便下次使用。
